approximate string searching
approximate string searching(approximate string matching)
馬馬虎虎的字串搜尋,只需大致符合,不必完全符合。
string distance
兩個字串的距離。字串差異程度。反過來就是字串相似程度。
經典的字串距離是edit distance(Levenshtein distance):新增、刪除、修改一個字元皆算做一步,一個字串變成另一個字串的最少步數。
演算法(dynamic time warping)
「longest common subsequence: dynamic programming」,比對成功、比對失敗不是加上1、0,而是加上自訂數值。
一、小心設定數值,讓計算結果是距離函數,以客觀衡量多個字串之間的差異程度,避免AB很像、BC很像、AC卻不像的情況。
二、自訂表格計算範圍,加快計算速度。例如只算索引值i j很接近的格子、不算數值太大太小的格子。
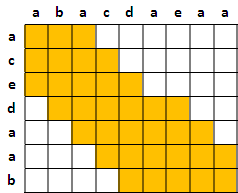
三、只取鄰近格子,有時太單純。多取幾格,設定代價。
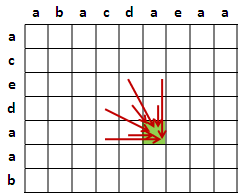
四、比對字元,有時太瑣碎。字串切成一段一段,以段為單位進行比對,變成兩層。
UVa 164 526 10739 12351
演算法(BK-tree)